晶体结构和冷冻电镜结构为计算机辅助药物设计提供了结构基础。然而,这些实验结构仅能提供静态水平的信息,而配体-蛋白、蛋白-蛋白的识别为动态过程,识别过程会经历很多重要的中间态。未来的药物设计不仅包括实验观察到的配体-受体结合状态,还包括整个配体-蛋白质识别途径中的各个中间态。中间态能够提供更详细的补充结构,能够拓展药物设计的新视角。配体-蛋白、蛋白-蛋白的识别发生在μs、ms甚至更长的时间尺度上,传统的分子动力学(cMD)方法难以在合理的时间内模拟到大规模的构象变化。因此,需要发展加速取样方法,提高模拟效率,在合理的模拟时间内捕捉到更多的动态信息。
近日,南开大学药学院、药物化学生物学国家重点实验室的林建平教授、李冬梅副教授团队在PNAS上发表了题为“The full activation mechanism of the adenosine A1 receptor revealed by GaMD and Su-GaMD simulations”的研究论文。研究团队开发了一种新的分子动力学方法——Su-GaMD,并揭示了腺苷(Ado)A1受体(A1R)的完全活化机制。

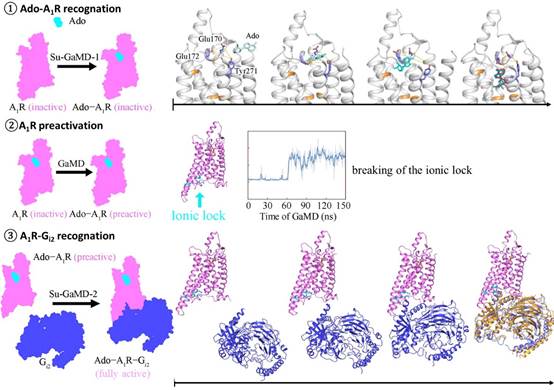
研究团队在标准的高斯加速分子动力学(GaMD)模拟中加入禁忌监督算法,开发了一种新的分子动力学方法——Su-GaMD。该方法能够在ns尺度揭示配体-蛋白、蛋白-蛋白的识别过程。模拟结果显示,A1R的完全活化包括三个阶段:Ado与A1R的识别、A1R的预活化、Gi2与A1R的识别。Ado的识别过程中,经历几个不同的中间态,识别路径中的Glu170ECL2、Phe171ECL2、Glu172ECL2, Asn2546.55、Thr2576.58、Lys265ECL3、Tyr2717.36和Thr2777.42等残基在前人的实验研究中均显示出对Ado的结合有重要作用。接着,Ado的结合引起A1R胞内端构象变化,Arg1053.50/Arg1083.53-Glu2296.30离子锁断开,A1R达到预活化状态。最后,Gi2在胞内端逐渐靠近A1R,通过ICLs(特别是ICL2)逐渐与A1R结合,形成与前人实验结构相一致的Ado-A1R-Gi2的复合物(Ado RMSD = 2.0 Å,A1R RMSD = 1.7 Å,Gαi RMSD = 2.9 Å)。A1R胞内端Gi2的结合使胞外端Ado结合口袋的体积缩小,增强了Ado与A1R的结合能力,这也与前人的β1肾上腺素能受体的实验现象相符合。
该研究在数百ns的模拟中实现了从游离的Ado、apo-A1R、Gi2到的Ado-A1R-Gi2复合物的动态全过程,揭示了A1R 完全活化的机理,这对于理解GPCR的信号转导机制至关重要。揭示的Ado和Gi2与A1R识别过程中的各个中间态拓展了A1R药物设计的新视角,为靶向A1R的药物研发提供了新思路。
南开大学药学院博士研究生李洋、孙霁雪为本论文的共同第一作者,李冬梅副教授和林建平教授为共同通讯作者。
论文链接:https://www.pnas.org/doi/10.1073/pnas.2203702119